Clasificación de cáncer cervical usando redes neuronales convolucionales, transferencia de aprendizaje y aumento de datos
Cervical cancer classification using convolutional neural networks, transfer learning and data augmentation

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-SinDerivadas 4.0.
Declaración del copyright
Los autores ceden en exclusiva a la Universidad EIA, con facultad de cesión a terceros, todos los derechos de explotación que deriven de los trabajos que sean aceptados para su publicación en la Revista EIA, así como en cualquier producto derivados de la misma y, en particular, los de reproducción, distribución, comunicación pública (incluida la puesta a disposición interactiva) y transformación (incluidas la adaptación, la modificación y, en su caso, la traducción), para todas las modalidades de explotación (a título enunciativo y no limitativo: en formato papel, electrónico, on-line, soporte informático o audiovisual, así como en cualquier otro formato, incluso con finalidad promocional o publicitaria y/o para la realización de productos derivados), para un ámbito territorial mundial y para toda la duración legal de los derechos prevista en el vigente texto difundido de la Ley de Propiedad Intelectual. Esta cesión la realizarán los autores sin derecho a ningún tipo de remuneración o indemnización.
La autorización conferida a la Revista EIA estará vigente a partir de la fecha en que se incluye en el volumen y número respectivo en el Sistema Open Journal Systems de la Revista EIA, así como en las diferentes bases e índices de datos en que se encuentra indexada la publicación.
Todos los contenidos de la Revista EIA, están publicados bajo la Licencia Creative Commons Atribución-NoComercial-NoDerivativa 4.0 Internacional
Mostrar biografía de los autores
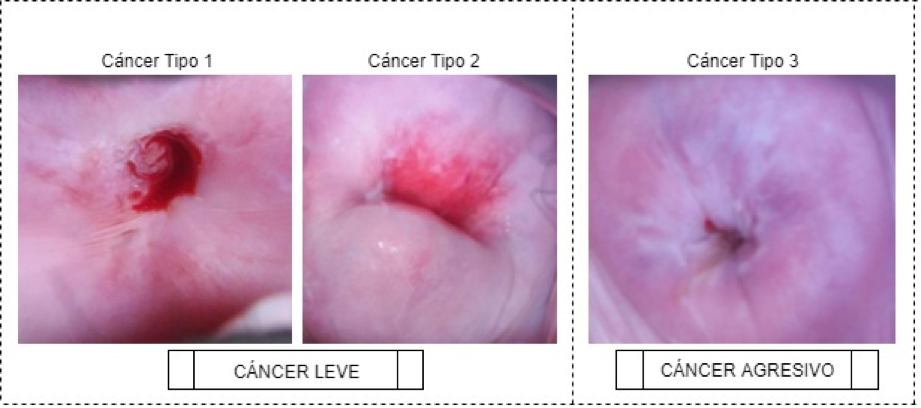
El cáncer cervical se forma en las células que revisten el cuello uterino y la parte inferior del útero. Debido a razones de costo y baja oferta de servicios destinados a la detección de este tipo de cáncer, muchas mujeres no tienen acceso a un diagnóstico pronto y preciso, ocasionando un inicio tardío del tratamiento. Para dar solución a este problema se implementó una metodología que clasifica de manera automática el tipo de cáncer cervical, entre leve (Tipo 1 y 2) y agresivo (Tipo 3), utilizando técnicas de procesamiento digital de imágenes y aprendizaje profundo. Se trabajó en la construcción de un modelo computacional con base en redes neuronales convolucionales, transferencia de aprendizaje y aumento de datos, obteniendo precisiones de clasificación de hasta 97,35% sobre los datos de validación, asegurando la confiabilidad de los resultados. Con este trabajo se demostró que el diseño propuesto puede ser usado como un complemento para mejorar la eficiencia de las herramientas del diagnóstico asistido del cáncer.
Visitas del artículo 1875 | Visitas PDF 877
Descargas
- McGuire S. World cancer report 2014. Geneva, Switzerland: World Health Organization, international agency for research on cancer, WHO Press, 2015. Advances in Nutrition: An International Review Journal, 7, 418-419, 2016.
- Akshaya R., Manie R., Monisha B., Ranichadra V. Convolutional Neural Networks Aiding Colposcopy Image Classification. International Journal of Trend in Research and development, 5, 270-274, 2018.
- Almonte M., Sánchez G.I., Jerónimo J., Ferreción C., Lazcano E., Herrera R. Nuevos Paradigmas en la Prevención y Control de Cáncer de Cuello Uterino en América Latina. Salud Pública de México, 52, No 6, 2010.
- Lorena M., Villate S., Jiménez D., Conduct in regard to the papanicolaou test: The voice of the patients in face of abnormal growth in the cervix, Revista Colombiana de Enfermería, Vol. 18, páginas 1-13, 2019
- Kaur N., Panagrahi N., Mittal A. Automated Cervical Cancer Screening Using Transfer Learning. International Journal Of Advanced Research in Science and Engineering, 6, 2110-2119, 2017.
- Intel & MobileODT, Cervical Cancer Screening, 2017, [Online]. Available: https://www.kaggle.com/c/intel-mobileodt-cervical-cancer-screening/data
- Simonyan K., Zisserman A. Very Deep Convolutional Networks for Large Scale Image Recognition. Published as a conference paper at ICLR 2015. San Diego, California, Estados Unidos, abril, 2015.
- Park Chansung, Transfer Learning in Tensorflow (VGG19 on CIFAR-10): Part 1, 2018, 10 Octubre 2019, [Online]. Available: https://towardsdatascience.com/transfer-learning-in-tensorflow-9e4f7eae3bb4
- Stanford University, Princeton University, ImageNet, 2016, 10 Octubre 2019, [Online]. Available: http://www.image-net.org/
- Zhang XQ, Zhao S-G, Cervical image classification based on image segmentation preprocessing and a CapsNet network model, Wiley, páginas 19-28, 2019 , [Online]. Available: https://doi.org/10.1002/ima.22291
- Fernandes K., Cardoso J., Fernandes J., Automated Methods for the Decision Support of Cervical Cancer Screening Using Digital Colposcopies, IEEE Xplore, Vol. 6, páginas 33910-33927, 2018, [Online]. Available: https://ieeexplore.ieee.org/document/8362613
- Vasudha, Mittal A., Juneja M., Cervix Cancer Classification using Colposcopy Images by Deep Learning Method, IJETSR, Vol. 5, páginas 426-432, 2018, [Online]. Available: https://pdfs.semanticscholar.org/f099/0cd17037129f7a55fcdf279ea6e9d613e8fe.pdf
- Caraiman S., Vasile I., Histogram-based segmentation of quantum images, ELSEVIER, Vol. 529, páginas 46-60, 2014, [Online]. Available: https://www.sciencedirect.com/science/article/pii/S0304397513005835
- Adrian Rosebrock, pyimagesearch, Keras ImageDataGenerator and Data Augmentation(Julio 8, 2019), consultado por última vez el 10 de octubre del 2019 en: https://www.pyimagesearch.com/2019/07/08/keras-imagedatagenerator-and-data-augmentation/?utm_source=facebook&utm_medium=ad-08-07-2019&utm_campaign=8+July+2019+BP+-+Traffic&utm_content=Default+name+-+Traffic&fbid_campaign=6116019415846&fbid_adset=6116019416246&utm_adset=1+July+2019+BP+-+All+Visitors+90+Days+-+Worldwide+-+18%2B&fbid_ad=6116019417246
- Mikolajczyk A. Grochowski M, Data augmentation for improving deep learning in image classification problem, IEEE Xplore, Poland, 2018, 21 Junio 2018, [Online]. Available: https://ieeexplore.ieee.org/document/8388338
- Intel & MobileODT, Cervical Cancer Screening, 2017, [Online]. Available: https://www.kaggle.com/c/intel-mobileodt-cervical-cancer-screening
- Tiago S. Nazar´e, Gabriel B. Paranhos da Costa, Welinton A. Contato, and Moacir Ponti, Deep Convolutional Neural Networks and Noisy Images, ResearchGate, paginas 416-424, 2018, [Online]. Available: https://www.researchgate.net/publication/322915518_Deep_Convolutional_Neural_Networks_and_Noisy_Images
- Nawal M. Nour, Cervical Cancer: A Preventable Death, Obstet Gynecol, Vol. 2, páginas 240-244, 2009, [Online]. Available: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2812875/
- Ayan E, H. Muray Ü, Data augmentation importance for classification of skin lesions via deep learning, IEEE Xplore, páginas 1-5, 2019, [Online]. Available: https://ieeexplore.ieee.org/document/8391469/citations?tabFilter=papers#citations
- Keras Documentation, Keras, [Online]. Available: https://keras.io/why-use-keras/
- . TensorFlow Core r1.14, Tensorflow, [Online]. Available: https://www.tensorflow.org/versions/r1.14/api_docs/python/tf
- Krizhevsky A., Sutskever I., Hinton G.E. ImageNet Classification with Deep Convolutional Neural Networks. In NIPS, 2012.
- Abien Fred M. Agarap, Cornell University, Deep Learning using Rectified Linear Units (ReLU), 2019, 7 febrero 2019, [Online]. Available: https://arxiv.org/abs/1803.08375
- Sridhar Narayan, The generalized sigmoid activation function: Competitive supervised learning, ScienceDirect, Vol. 99, páginas 69-82, 1997, [Online]. Available: https://www.sciencedirect.com/science/article/pii/S0020025596002009
- Daniel Godoy, Towards Data Science, Understanding binary cross-entropy / log loss: a visual explanation, 2018, 10 octubre 2019, [Online]. Available: https://towardsdatascience.com/understanding-binary-cross-entropy-log-loss-a-visual-explanation-a3ac6025181a
- Zhang S., Choromanska A., and LeCun Y.. Deep learning with Elastic Averaging SGD. Neural Information Processing Systems Conference (NIPS 2015), Vol. 28, páginas 1–24, 2015, [Online]. Available: https://papers.nips.cc/paper/5761-deep-learning-with-elastic-averaging-sgd
- Piotr Skalski, Towards Data Science, Preventing Deep Neural Network from Overfitting, 2018, 10 Octubre 2019, [Online]. Available: https://towardsdatascience.com/preventing-deep-neural-network-from-overfitting-953458db800a
- Srivastava N., Hinton G., Krizhevsky A., Sutskever I., Salakhutdinov R., Dropout: A Simple Way to Prevent Neural Networks from Overfitting, Journal of Machine Learning Research, Vol. 15, páginas. 1929-1958, 2014, [Online]. Available: http://jmlr.org/papers/v15/srivastava14a.html




